Modelagem GRU pode ajudar na compreensão e controle de doenças emergentes
Pesquisa publicada na RSBMT utiliza redes neurais baseadas em Gated Recurrent Unit para análise de sequências genômicas do SARS-CoV-2, SARS, MERS e Ebola
03/02/2025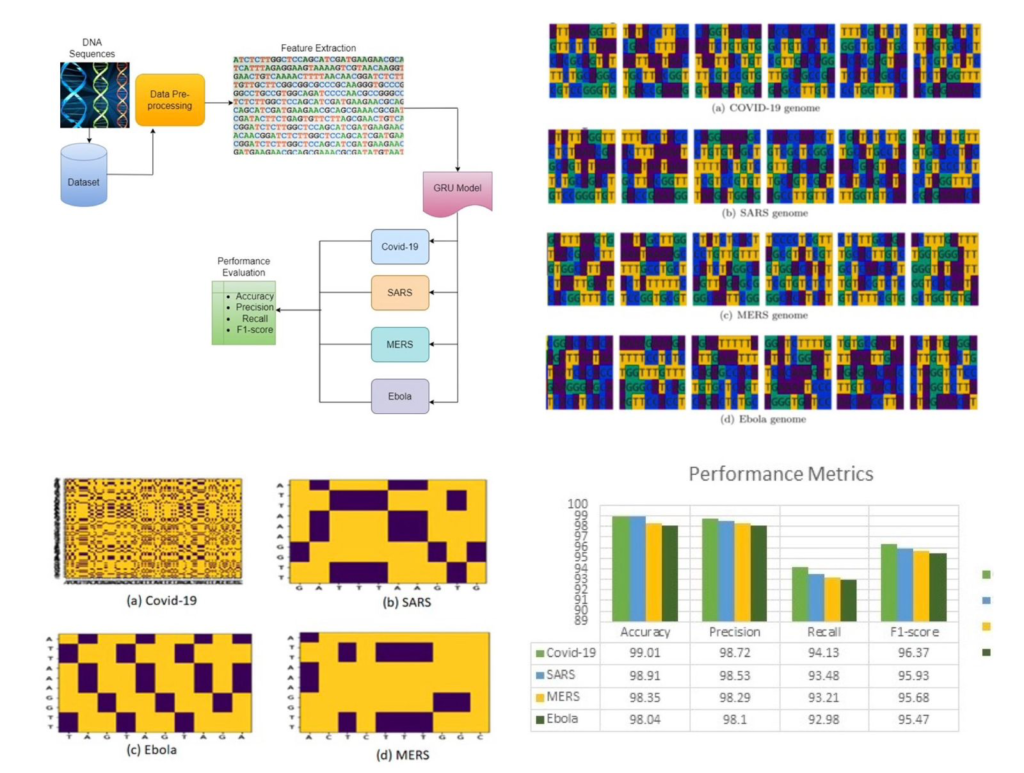
Nos últimos anos, epidemias como da Covid-19 evidenciaram a necessidade de tecnologias capazes de prever e mitigar crises sanitárias
As pandemias virais tornaram-se um dos maiores desafios globais do século XXI, e a velocidade de propagação de patógenos em um mundo altamente conectado exige soluções tecnológicas à altura. Neste contexto, uma pesquisa conduzida pelos doutores Abhishak Raj Devaraj e Victor Jose Marianthiran, publicada na Revista da Sociedade Brasileira de Medicina Tropical (RSBMT), trouxe contribuições significativas ao campo da genômica viral ao utilizar redes neurais baseadas em Gated Recurrent Unit (GRU, na sigla em inglês) para a análise de sequências genômicas de vírus como SARS-CoV-2, SARS, MERS e Ebola. Além de identificar padrões evolutivos com alta precisão, a abordagem abre novas perspectivas para o controle de surtos virais.
Desde a epidemia de SARS em 2003, o mundo tem enfrentado uma série de emergências de saúde pública, como os surtos de H1N1 e Zika, além da pandemia de Covid-19. De acordo com a publicação “Advancements in Viral Genomics: Gated Recurrent Unit Modeling of SARS-CoV-2, SARS, MERS, and Ebola viruses”, a crescente frequência e complexidade dessas emergências são impulsionadas por fatores como urbanização acelerada e aumento do tráfego global. Essas condições reforçam a necessidade de ferramentas que permitam não apenas reagir a surtos, mas também antecipar ameaças.
Os pesquisadores indianos desenvolveram um modelo para analisar sequências genômicas dos quatro vírus mais estudados na última década: SARS-CoV-2, SARS, MERS e Ebola. A pesquisa utilizou modelagem computacional para identificar padrões que possam orientar intervenções futuras. O estudo utilizou a GRU, um tipo de rede neural recorrente, especialmente eficaz na captura de dependências de curto e longo prazo em dados sequenciais. Essa capacidade é fundamental para a análise de genomas virais, que frequentemente apresentam padrões altamente complexos. Foram analisadas 1.920 sequências genômicas, convertidas em representações numéricas para alimentar o modelo. A GRU foi escolhida por sua capacidade superior de processar dados temporais e identificar variações genéticas entre diferentes cepas virais.
O estudo envolveu um rigoroso processo de pré-processamento dos dados, incluindo a eliminação de redundâncias genômicas, a conversão de DNA em RNA e a extração de atributos como conteúdo GC, padrões de códon, sequência de aminoácidos e informações estruturais. O modelo demonstrou alta eficácia na identificação de padrões genômicos, alcançando uma precisão de 99,01% para o SARS-CoV-2, 98,91% para SARS, 98,35% para MERS e 98,04% para Ebola. Além disso, os valores de F1-score variaram de 95,47% a 96,37%, indicando alta confiabilidade na classificação de genomas virais e no reconhecimento de variantes.
Para complementar a análise, os pesquisadores utilizaram gráficos de pontos para visualizar relações genéticas entre os vírus, revelando padrões importantes de similaridade e diferenças genômicas. Essas visualizações facilitaram a compreensão das conexões evolutivas e forneceram insights sobre como os vírus se adaptam e interagem com seus hospedeiros. A equipe também realizou análises filogenéticas para identificar mutações associadas à virulência e à transmissão. Como resultado, foi possível mapear hotspots de provável disseminação do Ebola e de outros vírus emergente, facilitando intervenções precoces.
Os achados demonstram que a modelagem GRU pode ser aplicada para prever mutações que aumentam a transmissibilidade ou virulência dos vírus, além de auxiliar no desenvolvimento de ferramentas de diagnóstico e terapias personalizadas. O estudo também evidenciou que o uso de dot plots pode ilustrar com precisão as diferenças e similaridades entre os genomas analisados, contribuindo para o aprimoramento das estratégias de contenção.
Embora os resultados sejam promissores, os autores reconhecem desafios, como a dependência de dados genômicos de alta qualidade. Sequências incompletas ou ruidosas podem comprometer a análise. Além disso, o alto custo computacional para processar grandes volumes de dados representa um obstáculo significativo. Para este estudo, os pesquisadores utilizaram dados genômicos disponíveis no banco público GenBank, baixados em 4 de março de 2024. Segundo os autores, avanços na eficiência dos algoritmos são essenciais para permitir análise em escala global. Eles também ressaltam a importância de colaborações multidisciplinares entre bioinformatas, cientistas de dados e biólogos para ampliar o impacto da modelagem computacional no combate a doenças infecciosas.
A aplicação da GRU na análise genômica representa um avanço significativo na compreensão de vírus emergentes. O modelo não apenas amplia o conhecimento sobre a dinâmica evolutiva desses patógenos, mas também oferece ferramentas práticas para diagnóstico e controle de surtos. A pesquisa pulicada na RSBMT reforça o papel da inteligência artificial como aliada da saúde pública, trazendo perspectivas promissoras para um futuro mais preparado diante de novas ameaças virais.
A equipe pretende expandir a aplicação da GRU para analisar outros vírus emergentes, como Nipah e Marburg, além de integrar dados ômicos complementares, como proteômica e transcriptômica. O objetivo é desenvolver um sistema de vigilância genômica capaz de detectar e responder rapidamente a novas ameaças virais. Ao combinar rigor científico e inovação tecnológica, este estudo representa um marco na luta contra doenças emergentes e destaca relevância da ciência no enfrentamento dos desafios de um mundo interconectado.
Confira o artigo “Advancements in Viral Genomics: Gated Recurrent Unit Modeling of SARS-CoV-2, SARS, MERS, and Ebola viruses” publicado na Revista da Sociedade Brasileira de Medicina Tropical (RSBMT): https://doi.org/10.1590/0037-8682-0178-2024.
**Esta reportagem reflete exclusivamente a opinião do entrevistado.**